열심히 코딩 하숭!
타이타닉 생존자 예측, 분류 성능 평가 지표(Accuracy, Confusion Matrix, Precision, Recall, F1 Score, ROC-AUC), 피마 인디언 당뇨병 예측 | 3주차 - 3, 4 | 파이썬 머신러닝 완벽가이드 본문
타이타닉 생존자 예측, 분류 성능 평가 지표(Accuracy, Confusion Matrix, Precision, Recall, F1 Score, ROC-AUC), 피마 인디언 당뇨병 예측 | 3주차 - 3, 4 | 파이썬 머신러닝 완벽가이드
채숭이 2023. 1. 22. 16:56* 해당 글은 inflearn의 강의 '[개정판] 파이썬 머신러닝 완벽가이드'를 정리한 글입니다.
회색 - 강의 제목
노란색, 주황색 - 강조
3일
사이킷런으로 수행하는 타이타닉 생존자 예측
타이타닉 생존자 예측 - 데이터 전처리
- Null 처리 / 불필요한 속성 제거 / 인코딩
타이타닉 생존자 예측 - 모델 학습 및 검증/예측/평가
- 결정트리, 랜덤포레스트, 로지스틱 회귀 학습 비교
- K 폴드 교차 검증
- cross_val_score()와 GridSearchCV() 수행
코드
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
titanic_df = pd.read_csv('./titanic_train.csv')
titanic_df.head(3)결과

- Passengerid: 탑승자 데이터 일련번호
- survived: 생존 여부, 0 = 사망, 1 = 생존
- Pclass: 티켓의 선실 등급, 1 = 일등석, 2 = 이등석, 3 = 삼등석
- sex: 탑승자 성별
- name: 탑승자 이름
- Age: 탑승자 나이
- sibsp: 같이 탑승한 형제자매 또는 배우자 인원수
- parch: 같이 탑승한 부모님 또는 어린이 인원수
- ticket: 티켓 번호
- fare: 요금
- cabin: 선실 번호
- embarked: 중간 정착 항구 C = Cherbourg, Q = Queenstown, S = Southampton
-----
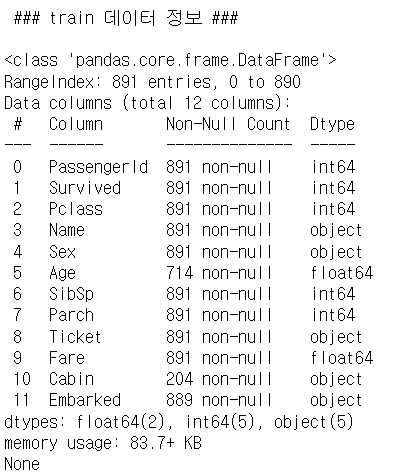
print('\n ### train 데이터 정보 ### \n')
print(titanic_df.info())
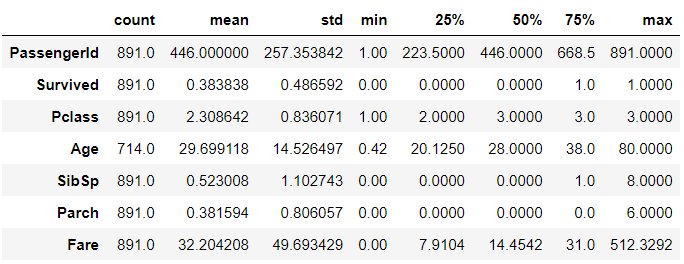
#titanic_df.describe()
titanic_df.describe().transpose() #행과 열 바꿈결과
-----
NULL 컬럼들에 대한 처리
titanic_df['Age'].fillna(titanic_df['Age'].mean(), inplace=True)
titanic_df['Cabin'].fillna('N', inplace=True) # 아예 새로운 값인 'N'으로 대체
titanic_df['Embarked'].fillna('N', inplace=True) # 아예 새로운 값인 'N'으로 대체
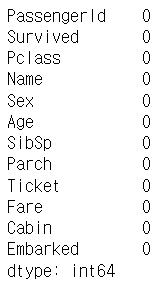
print('데이터 세트 Null 값 갯수 ',titanic_df.isnull().sum().sum())결과
데이터 세트 Null 값 갯수 0
-----
titanic_df.isnull().sum()결과
-----
주요 컬럼 EDA (=Exploratory Data Analysis, 수집한 데이터를 다각도로 관찰하고 이해하는 과정)
object 컬럼 추출
# object 컬럼타입 추출
titanic_df.dtypes[titanic_df.dtypes == 'object'].index.tolist()결과
['Name', 'Sex', 'Ticket', 'Cabin', 'Embarked']
-----
분포 확인
print(' Sex 값 분포 :\n',titanic_df['Sex'].value_counts())
print('\n Cabin 값 분포 :\n',titanic_df['Cabin'].value_counts())
print('\n Embarked 값 분포 :\n',titanic_df['Embarked'].value_counts()) Sex 값 분포 :
male 577
female 314
Name: Sex, dtype: int64
Cabin 값 분포 :
N 687
C23 C25 C27 4
G6 4
B96 B98 4
C22 C26 3
...
E34 1
C7 1
C54 1
E36 1
C148 1
Name: Cabin, Length: 148, dtype: int64
Embarked 값 분포 :
S 644
C 168
Q 77
N 2
Name: Embarked, dtype: int64
-----
문자열 데이터의 첫 글자만 가져옴
titanic_df['Cabin'] = titanic_df['Cabin'].str[:1]
print(titanic_df['Cabin'].head(3))결과
-----
위 결과 확인
titanic_df['Cabin'].value_counts()결과
N 687
C 59
B 47
D 33
E 32
A 15
F 13
G 4
T 1
Name: Cabin, dtype: int64
groupby한 후 특정 column에 대해 count
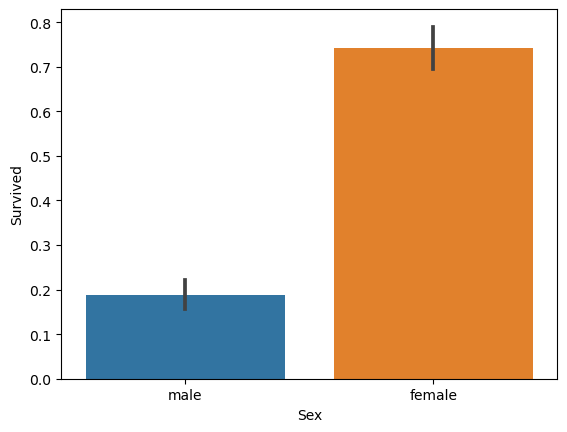
titanic_df.groupby(['Sex','Survived'])['Survived'].count() # groupby한 후 특정 column에 대해 count결과
Sex Survived
female 0 81
1 233
male 0 468
1 109
Name: Survived, dtype: int64
그래프로 나타내기
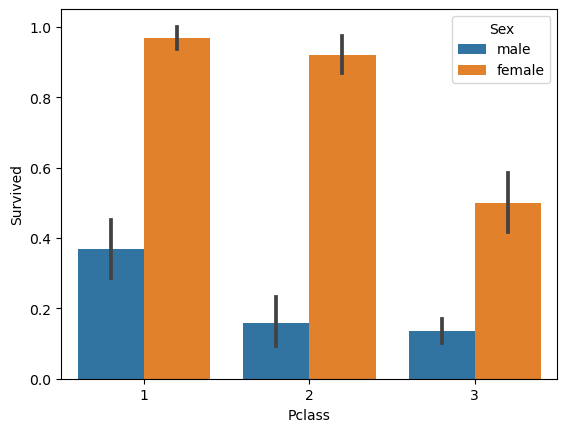
sns.barplot(x='Sex', y = 'Survived', data=titanic_df) # Survived는 0 또는 1이므로 y축의 범위가 0 ~ 1임sns.barplot(x='Pclass', y='Survived', hue='Sex', data=titanic_df) # hue를 사용하여 속성 추가결과
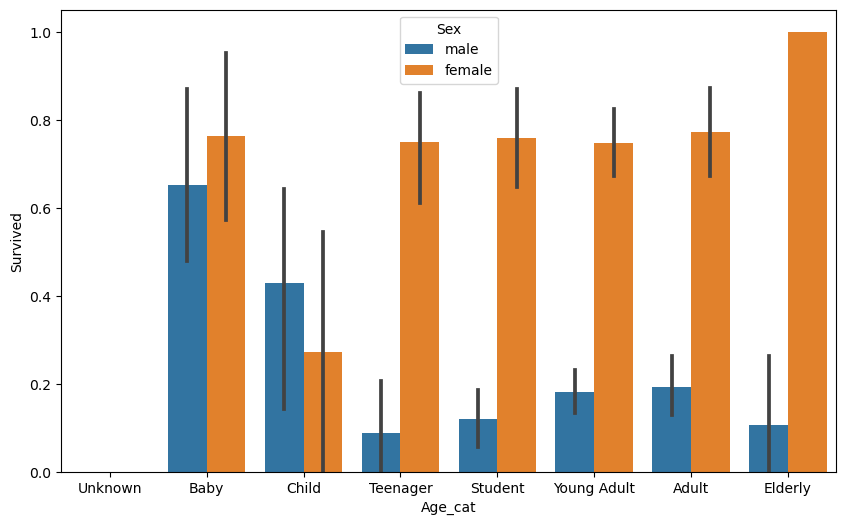
입력 age에 따라 구분값을 반환하는 함수 설정 - apply lambda
# 입력 age에 따라 구분값을 반환하는 함수 설정. DataFrame의 apply lambda식에 사용.
def get_category(age):
cat = ''
if age <= -1: cat = 'Unknown'
elif age <= 5: cat = 'Baby'
elif age <= 12: cat = 'Child'
elif age <= 18: cat = 'Teenager'
elif age <= 25: cat = 'Student'
elif age <= 35: cat = 'Young Adult'
elif age <= 60: cat = 'Adult'
else: cat = 'Elderly'
return cat
# 막대그래프의 크기 figure를 더 크게 설정
plt.figure(figsize=(10,6))
#X축의 값을 순차적으로 표시하기 위한 설정
group_names = ['Unknown', 'Baby', 'Child', 'Teenager', 'Student', 'Young Adult', 'Adult', 'Elderly']
# lambda 식에 위에서 생성한 get_category( ) 함수를 반환값으로 지정.
# get_category(X)는 입력값으로 'Age' 컬럼값을 받아서 해당하는 cat 반환
# 새로운 속성 만들기
titanic_df['Age_cat'] = titanic_df['Age'].apply(lambda x : get_category(x))
sns.barplot(x='Age_cat', y = 'Survived', hue='Sex', data=titanic_df, order=group_names)
titanic_df.drop('Age_cat', axis=1, inplace=True)결과
인코딩
from sklearn.preprocessing import LabelEncoder
def encode_features(dataDF):
features = ['Cabin', 'Sex', 'Embarked']
le = LabelEncoder()
for feature in features:
le.fit(dataDF[feature])
dataDF[feature] = le.transform(dataDF[feature])
return dataDF
titanic_df = encode_features(titanic_df)
titanic_df.head()결과
전처리 모아보기
from sklearn.preprocessing import LabelEncoder
# Null 처리 함수
def fillna(df):
df['Age'].fillna(df['Age'].mean(),inplace=True)
df['Cabin'].fillna('N',inplace=True)
df['Embarked'].fillna('N',inplace=True)
return df
# 머신러닝 알고리즘에 불필요한 피처 제거
def drop_features(df):
df.drop(['PassengerId','Name','Ticket'], axis=1, inplace=True)
return df
# 레이블 인코딩 수행.
def format_features(df):
df['Cabin'] = df['Cabin'].str[:1]
features = ['Cabin','Sex','Embarked']
for feature in features:
le = LabelEncoder()
le = le.fit(df[feature])
df[feature] = le.transform(df[feature])
return df
# 앞에서 설정한 Data Preprocessing 함수 호출
def transform_features(df):
df = fillna(df)
df = drop_features(df)
df = format_features(df)
return df
원본데이터 재로딩 & feature와 label 데이터 구분하여 추출
# 원본 데이터를 재로딩 하고, feature데이터 셋과 Label 데이터 셋 추출.
titanic_df = pd.read_csv('./titanic_train.csv')
y_titanic_df = titanic_df['Survived']
X_titanic_df= titanic_df.drop('Survived',axis=1, inplace=False)
X_titanic_df = transform_features(X_titanic_df)
train_test_split 함수
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test=train_test_split(X_titanic_df, y_titanic_df, \
test_size=0.2, random_state=11)
print(X_train.shape, X_test.shape, y_train.shape, y_test.shape)결과
(712, 8) (179, 8) (712,) (179,)
결정트리, Random Forest, 로지스틱 회귀 - 사이킷런 Classifier 클래스
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
# 결정트리, Random Forest, 로지스틱 회귀를 위한 사이킷런 Classifier 클래스 생성
dt_clf = DecisionTreeClassifier(random_state=11)
rf_clf = RandomForestClassifier(random_state=11)
lr_clf = LogisticRegression(solver='liblinear')
# DecisionTreeClassifier 학습/예측/평가
dt_clf.fit(X_train , y_train)
dt_pred = dt_clf.predict(X_test)
print('DecisionTreeClassifier 정확도: {0:.4f}'.format(accuracy_score(y_test, dt_pred)))
# RandomForestClassifier 학습/예측/평가
rf_clf.fit(X_train , y_train)
rf_pred = rf_clf.predict(X_test)
print('RandomForestClassifier 정확도:{0:.4f}'.format(accuracy_score(y_test, rf_pred)))
# LogisticRegression 학습/예측/평가
lr_clf.fit(X_train , y_train)
lr_pred = lr_clf.predict(X_test)
print('LogisticRegression 정확도: {0:.4f}'.format(accuracy_score(y_test, lr_pred)))결과
DecisionTreeClassifier 정확도: 0.7877
RandomForestClassifier 정확도:0.8547
LogisticRegression 정확도: 0.8659
K fold 검증 - 직접 구하는 코드
from sklearn.model_selection import KFold
def exec_kfold(clf, folds=5):
# 폴드 세트를 5개인 KFold객체를 생성, 폴드 수만큼 예측결과 저장을 위한 리스트 객체 생성.
kfold = KFold(n_splits=folds)
scores = []
# KFold 교차 검증 수행.
for iter_count , (train_index, test_index) in enumerate(kfold.split(X_titanic_df)):
# X_titanic_df 데이터에서 교차 검증별로 학습과 검증 데이터를 가리키는 index 생성
# values: ndarray로 바뀜
X_train, X_test = X_titanic_df.values[train_index], X_titanic_df.values[test_index]
y_train, y_test = y_titanic_df.values[train_index], y_titanic_df.values[test_index]
# Classifier 학습, 예측, 정확도 계산
clf.fit(X_train, y_train)
predictions = clf.predict(X_test)
accuracy = accuracy_score(y_test, predictions)
scores.append(accuracy)
print("교차 검증 {0} 정확도: {1:.4f}".format(iter_count, accuracy))
# 5개 fold에서의 평균 정확도 계산.
mean_score = np.mean(scores)
print("평균 정확도: {0:.4f}".format(mean_score))
# exec_kfold 호출
exec_kfold(dt_clf , folds=5)결과
교차 검증 0 정확도: 0.7542
교차 검증 1 정확도: 0.7809
교차 검증 2 정확도: 0.7865
교차 검증 3 정확도: 0.7697
교차 검증 4 정확도: 0.8202
평균 정확도: 0.7823
K fold 검증 - cross_val_score 함수
from sklearn.model_selection import cross_val_score
scores = cross_val_score(dt_clf, X_titanic_df , y_titanic_df , cv=5)
for iter_count,accuracy in enumerate(scores):
print("교차 검증 {0} 정확도: {1:.4f}".format(iter_count, accuracy))
print("평균 정확도: {0:.4f}".format(np.mean(scores)))결과
교차 검증 0 정확도: 0.7430
교차 검증 1 정확도: 0.7753
교차 검증 2 정확도: 0.7921
교차 검증 3 정확도: 0.7865
교차 검증 4 정확도: 0.8427
평균 정확도: 0.7879
GridSearchCV 코드
from sklearn.model_selection import GridSearchCV
parameters = {'max_depth':[2,3,5,10],
'min_samples_split':[2,3,5], 'min_samples_leaf':[1,5,8]}
grid_dclf = GridSearchCV(dt_clf, param_grid=parameters, scoring='accuracy', cv=5)
grid_dclf.fit(X_train, y_train)
print('GridSearchCV 최적 하이퍼 파라미터 :', grid_dclf.best_params_)
print('GridSearchCV 최고 정확도: {0:.4f}'.format(grid_dclf.best_score_))
best_dclf = grid_dclf.best_estimator_
# GridSearchCV의 최적 하이퍼 파라미터로 학습된 Estimator로 예측 및 평가 수행.
dpredictions = best_dclf.predict(X_test)
accuracy = accuracy_score(y_test , dpredictions)
print('테스트 세트에서의 DecisionTreeClassifier 정확도 : {0:.4f}'.format(accuracy))결과
GridSearchCV 최적 하이퍼 파라미터 : {'max_depth': 3, 'min_samples_leaf': 5, 'min_samples_split': 2}
GridSearchCV 최고 정확도: 0.7992
테스트 세트에서의 DecisionTreeClassifier 정확도 : 0.8715
분류(Classification) 성능 평가지표 개요와 정확도(Accuracy) 소개
분류(Classification) 성능 평가 지표
- 정확도(Accuracy)
- 오차행렬(Confusion Matrix)
- 정밀도(Precision)
- F1 스코어
- ROC AUC
1) 정확도(Accuracy)

- 직관적으로 모델 예측 성능을 나타내는 평가 지표
- 문제점1: 이진 분류의 경우 데이터의 구성에 따라 ML 모델의 성능을 왜곡할 수 있기 때문에
정확도 하나만을 가지고 모든 성능을 평가하는 것은 옳지 않음
- 문제점2: 정확도는 불균형한(imbalanced) 레이블 값 분포에서 ML 모델의 성능을 판단할 경우 적합한 지표가 아님
(대충 이런 느낌이다~)
from sklearn.metrics import accuracy_score
X_train, X_test, y_train, y_test=train_test_split(X_titanic_df, y_titanic_df, \
test_size=0.2, random_state=0)
# (생략->) 위에서 생성한 Dummy Classifier를 이용하여 학습/예측/평가 수행
myclf = MyDummyClassifier()
myclf.fit(X_train ,y_train)
mypredictions = myclf.predict(X_test)
print('Dummy Classifier의 정확도는: {0:.4f}'.format(accuracy_score(y_test , mypredictions)))결과
Dummy Classifier의 정확도는: 0.7877
[문제점과 관련된 예제가 있는데, 중요한 건 아난 것 같아서 PASS 하겠음
> 3.1 정확도(Accuracy) ~ 3-5_ROC_AUC까지 예제 < 파일 참고]
오차행렬(Confusion Matrix), 정밀도(Precision), 재현율(Recall) 소개
2) 오차행렬(Confusion Matrix)

- 이진분류의 예측 오류가 얼마인지와 더불어, 어떠한 유형의 예측 오류가 발생하고 있는지를 함께 나타내는 지표
- True Negative의 경우) Negative로 예측했는데 실제 그 예측값이 맞을 경우(=True)
from sklearn.metrics import confusion_matrix
# 앞절의 예측 결과인 fakepred와 실제 결과인 y_test의 Confusion Matrix출력
confusion_matrix(y_test , fakepred) # 실제 먼저, 예측 나중결과
array([[405, 0],
[ 45, 0]], dtype=int64)TN FP
FN TP
→ 각각의 개수를 나타냄!
→ 여기에서는 FN이 45개이므로, 실제는 1인데 0으로 예측한 결과가 45건이 있다는 말임
→ 다 N쪽에 쏠려있으므로, 모델이 모든 예측을 0으로 한다는 것에 의구심을 품어야 함
- 정확도 = (예측 결과와 실제 값이 동일한 건수) / (전체 데이터 수) = (TN + TP) / (TN + FP + FN + TP)
3) 정밀도(Precision)와 재현율(Recall)
- 정밀도(Precision) = TP / (FP + TP)
- 재현율(Recall) = TP / (FN + TP)
- 정밀도 : 예측을 Positive(1)로 한 대상 중에 예측과 실제 값이 일치한 비율
- 재현율 : 실제 값이 Positive(1)인 대상 중에 예측과 실제 값이 일치한 비율
→ 둘다 이진 분류에서 많이 쓰임
→ 특히, 재현율은 의학계, 헬스케어 쪽에서 많이 쓰임
코드
from sklearn.metrics import accuracy_score, precision_score , recall_score
print("정밀도:", precision_score(y_test, fakepred))
print("재현율:", recall_score(y_test, fakepred))
-----
오차행렬, 정확도, 정밀도, 재현율을 한꺼번에 계산하는 함수 생성
from sklearn.metrics import accuracy_score, precision_score , recall_score , confusion_matrix
def get_clf_eval(y_test , pred):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
print('오차 행렬')
print(confusion)
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f}'.format(accuracy , precision ,recall))
-----
import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
import warnings
warnings.filterwarnings('ignore')
# 원본 데이터를 재로딩, 데이터 가공, 학습데이터/테스트 데이터 분할.
titanic_df = pd.read_csv('./titanic_train.csv')
y_titanic_df = titanic_df['Survived']
X_titanic_df= titanic_df.drop('Survived', axis=1)
X_titanic_df = transform_features(X_titanic_df)
X_train, X_test, y_train, y_test = train_test_split(X_titanic_df, y_titanic_df, \
test_size=0.20, random_state=11)
lr_clf = LogisticRegression(solver='liblinear')
lr_clf.fit(X_train , y_train)
pred = lr_clf.predict(X_test)
get_clf_eval(y_test , pred)결과
오차 행렬
[[108 10]
[ 14 47]]
정확도: 0.8659, 정밀도: 0.8246, 재현율: 0.7705
4일
정밀도와 재현율의 트레이드오프(Trade off)
업무에 따른 재현율과 정밀도의 상대적 중요도
- 재현율: 실제 Positive(양성)인 데이터 예측을 Negative(음성)로 잘못 판단하게 되면 큰 영향이 발생
ex) 암 진단, 금융사기 판별
- 정밀도: 실제 Negative(음성)인 데이터 예측을 Positive(양성)로 잘못 판단하게 되면 큰 영향 발생
ex) 스팸 메일
※ 불균형한 레이블 클래스를 가지는 이진 분류 모델에서는, 보통 중점적으로 찾아야하는 매우 적은 수의 결과값에 Positive를 설정해 값 1을 부여하고, 그렇지 않은 경우에는 Negative로 값 0을 부여
정밀도/재현율 트레이드 오프(Trade-off)
- 분류의 결정 임곗값(Threshold)을 조정해 정밀도 또는 재현율의 수치를 높일 수 있음
- But, 정밀도와 재현율은 상호 보완적인 평가 지표이지 때문에, 어느 한쪽을 강제로 높이면 다른 하나의 수치는 떨어지기 쉬움
→ 정밀도/재현율 트레이드 오프(Trade-off)
분류 결정 임곗값(Threshold)
- 확률의 기준을 결정함
- 기준에 따라 임곗값이 0.4일 경우, 0.4 이상의 확률을 가진 모든 데이터는 Positive로 판별됨
- 분류 결정 임곗값이 낮아질 수록 Positive로 예측할 확률이 높아짐. (→ 재현율 증가)

분류 결정 임곗값사용
- 사잇킷런 Estimator 객체의 predict_proba() 메소드는 분류 결정 예측 확률을 반환함
- 이를 이용하여 임의로 분류 결정 임곗값을 조정하면서 예측 확률을 변경할 수 있다
- 사이킷런의 precision_recall_curve() 함수를 통해 임곗값에 따른 정밀도, 재현율의 변화값을 제공
predict_proba( ) 메소드 확인
- 0이 될 확률과 1이 될 확률을 추출해줌
pred_proba = lr_clf.predict_proba(X_test)
pred = lr_clf.predict(X_test)
print('pred_proba()결과 Shape : {0}'.format(pred_proba.shape))
print('pred_proba array에서 앞 3개만 샘플로 추출 \n:', pred_proba[:3])
# -> 하나의 데이터에 대해 0이 될 확률과 1이 될 확률을 추출해줌
# -> 왼쪽과 오른쪽 중에 더 큰 값 쪽의 결과를 반환함
# 예측 확률 array 와 예측 결과값 array 를 concatenate 하여 예측 확률과 결과값을 한눈에 확인
pred_proba_result = np.concatenate([pred_proba , pred.reshape(-1,1)],axis=1)
print('두개의 class 중에서 더 큰 확률을 클래스 값으로 예측 \n',pred_proba_result[:3])결과
pred_proba()결과 Shape : (179, 2)
pred_proba array에서 앞 3개만 샘플로 추출
: [[0.44935227 0.55064773]
[0.86335512 0.13664488]
[0.86429645 0.13570355]]
두개의 class 중에서 더 큰 확률을 클래스 값으로 예측
[[0.44935227 0.55064773 1. ]
[0.86335512 0.13664488 0. ]
[0.86429645 0.13570355 0. ]]
Binarizer 활용
- threshold 기준값보다 같거나 작으면 0, 크면 1 반환
from sklearn.preprocessing import Binarizer
X = [[ 1, -1, 2],
[ 2, 0, 0],
[ 0, 1.1, 1.2]]
# threshold 기준값보다 같거나 작으면 0을, 크면 1을 반환
binarizer = Binarizer(threshold=1.1)
print(binarizer.fit_transform(X))결과
[[0. 0. 1.]
[1. 0. 0.]
[0. 0. 1.]]
분류 결정 임계값 0.4 기반에서 Binarizer를 이용하여 예측값 변환
# Predict 함수 흉내내기
from sklearn.preprocessing import Binarizer
# Binarizer의 threshold 설정값을 0.4로 설정. 즉 분류 결정 임곗값을 0.5에서 0.4로 낮춤
custom_threshold = 0.4
# predict_proba( ) 반환값의 두번째 컬럼 , 즉 Positive 클래스 컬럼 하나만 추출하여 Binarizer를 적용
pred_proba_1 = pred_proba[:,1].reshape(-1,1)
binarizer = Binarizer(threshold=custom_threshold).fit(pred_proba_1)
custom_predict = binarizer.transform(pred_proba_1)
get_clf_eval(y_test , custom_predict)결과
오차 행렬
[[97 21]
[11 50]]
정확도: 0.8212, 정밀도: 0.7042, 재현율: 0.8197
-> 여기에서 get_clf_eval은 전에 만들어뒀던 함수인데, 아래와 같음
- 오차행렬, 정확도, 정밀도, 재현율을 한꺼번에 계산해주는 함수임
from sklearn.metrics import accuracy_score, precision_score , recall_score , confusion_matrix
def get_clf_eval(y_test , pred):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
print('오차 행렬')
print(confusion)
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f}'.format(accuracy , precision ,recall))
여러개의 분류 결정 임곗값을 변경하면서 Binarizer를 이용하여 예측값 변환
# 테스트를 수행할 모든 임곗값을 리스트 객체로 저장.
thresholds = [0.4, 0.45, 0.50, 0.55, 0.60]
def get_eval_by_threshold(y_test , pred_proba_c1, thresholds):
# thresholds list객체내의 값을 차례로 iteration하면서 Evaluation 수행.
for custom_threshold in thresholds:
binarizer = Binarizer(threshold=custom_threshold).fit(pred_proba_c1)
custom_predict = binarizer.transform(pred_proba_c1)
print('임곗값:',custom_threshold)
get_clf_eval(y_test , custom_predict)
get_eval_by_threshold(y_test ,pred_proba[:,1].reshape(-1,1), thresholds )결과
임곗값: 0.4
오차 행렬
[[97 21]
[11 50]]
정확도: 0.8212, 정밀도: 0.7042, 재현율: 0.8197
임곗값: 0.45
오차 행렬
[[105 13]
[ 13 48]]
정확도: 0.8547, 정밀도: 0.7869, 재현율: 0.7869
임곗값: 0.5
오차 행렬
[[108 10]
[ 14 47]]
정확도: 0.8659, 정밀도: 0.8246, 재현율: 0.7705
임곗값: 0.55
오차 행렬
[[111 7]
[ 16 45]]
정확도: 0.8715, 정밀도: 0.8654, 재현율: 0.7377
임곗값: 0.6
오차 행렬
[[113 5]
[ 17 44]]
정확도: 0.8771, 정밀도: 0.8980, 재현율: 0.7213
precision_recall_curve( ) 를 이용하여 임곗값에 따른 정밀도-재현율 값 추출
from sklearn.metrics import precision_recall_curve
# 레이블 값이 1일때의 예측 확률을 추출
pred_proba_class1 = lr_clf.predict_proba(X_test)[:, 1]
# 실제값 데이터 셋과 레이블 값이 1일 때의 예측 확률을 precision_recall_curve 인자로 입력
precisions, recalls, thresholds = precision_recall_curve(y_test, pred_proba_class1 )
print('반환된 분류 결정 임곗값 배열의 Shape:', thresholds.shape)
print('반환된 precisions 배열의 Shape:', precisions.shape)
print('반환된 recalls 배열의 Shape:', recalls.shape)
print('thresholds 5 sample:', thresholds[:5])
print('precisions 5 sample:', precisions[:5])
print('recalls 5 sample:', recalls[:5])
#반환된 임계값 배열 로우가 147건이므로 샘플로 10건만 추출하되, 임곗값을 15 Step으로 추출.
thr_index = np.arange(0, thresholds.shape[0], 15)
print('샘플 추출을 위한 임계값 배열의 index 10개:', thr_index)
print('샘플용 10개의 임곗값: ', np.round(thresholds[thr_index], 2))
# 15 step 단위로 추출된 임계값에 따른 정밀도와 재현율 값
print('샘플 임계값별 정밀도: ', np.round(precisions[thr_index], 3))
print('샘플 임계값별 재현율: ', np.round(recalls[thr_index], 3))결과
반환된 분류 결정 임곗값 배열의 Shape: (147,)
반환된 precisions 배열의 Shape: (148,)
반환된 recalls 배열의 Shape: (148,)
thresholds 5 sample: [0.11573101 0.11636721 0.11819211 0.12102773 0.12349478]
precisions 5 sample: [0.37888199 0.375 0.37735849 0.37974684 0.38216561]
recalls 5 sample: [1. 0.98360656 0.98360656 0.98360656 0.98360656]
샘플 추출을 위한 임계값 배열의 index 10개: [ 0 15 30 45 60 75 90 105 120 135]
샘플용 10개의 임곗값: [0.12 0.13 0.15 0.17 0.26 0.38 0.49 0.63 0.76 0.9 ]
샘플 임계값별 정밀도: [0.379 0.424 0.455 0.519 0.618 0.676 0.797 0.93 0.964 1. ]
샘플 임계값별 재현율: [1. 0.967 0.902 0.902 0.902 0.82 0.77 0.656 0.443 0.213]
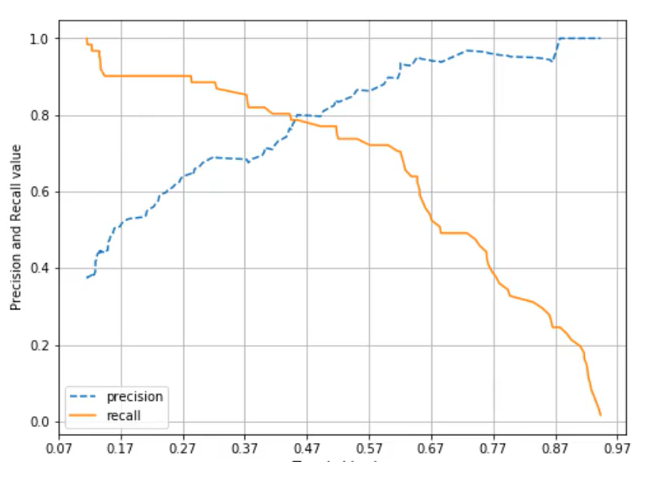
임곗값의 변경에 따른 정밀도-재현율 변화 곡선을 그림
import matplotlib.pyplot as plt
import matplotlib.ticker as ticker
%matplotlib inline
def precision_recall_curve_plot(y_test , pred_proba_c1):
# threshold ndarray와 이 threshold에 따른 정밀도, 재현율 ndarray 추출.
precisions, recalls, thresholds = precision_recall_curve( y_test, pred_proba_c1)
# X축을 threshold값으로, Y축은 정밀도, 재현율 값으로 각각 Plot 수행. 정밀도는 점선으로 표시
plt.figure(figsize=(8,6))
threshold_boundary = thresholds.shape[0]
plt.plot(thresholds, precisions[0:threshold_boundary], linestyle='--', label='precision')
plt.plot(thresholds, recalls[0:threshold_boundary],label='recall')
# -> [0:threshold_boundary]: threshold와의 개수를 맞춰주기 위해
# threshold 값 X 축의 Scale을 0.1 단위로 변경
start, end = plt.xlim()
plt.xticks(np.round(np.arange(start, end, 0.1),2))
# x축, y축 label과 legend, 그리고 grid 설정
plt.xlabel('Threshold value'); plt.ylabel('Precision and Recall value')
plt.legend(); plt.grid()
plt.show()
precision_recall_curve_plot( y_test, lr_clf.predict_proba(X_test)[:, 1] )결과

정밀도와 재현율의 맹점(모순되는 점)
- 정밀도를 100%로 만드는 방법: 확실한 기준이 되는 경우만 Positive로 예측하고 나머지는 모두 Negative로 예측
ex) 전체 환자 1000명 중 확실한 Positive 징후를 가진 환자 단 1명만 Positive로 예측하고 나머지는 모두 Negative로 예측해도 FP는 0, TP는 1이 되므로, 정밀도 = TP / (TP + FP) = 1 / (1+0) = 1 => 100%가 됨
- 재현율을 100%로 만드는 방법: 모든 환자를 Positive로 예측하면 됨
ex) 전체 환자 100명 중에 실제 양성인 사람이 30명 정도 있다고 할 때, 모두 Positive로 예측하게 되면, TP = 30, FN = 0이 되므로, 재현율 = TP / (TP + FN) = 30 / (30+0) = 1 = 100%가 됨
F1 Score와 ROC-AUC 이해
F1 Score
- 정밀도와 재현율을 결합한 지표
- 어느 한쪽으로 치우치지 않는 수치를 나타낼 때 상대적으로 높은 값을 가짐
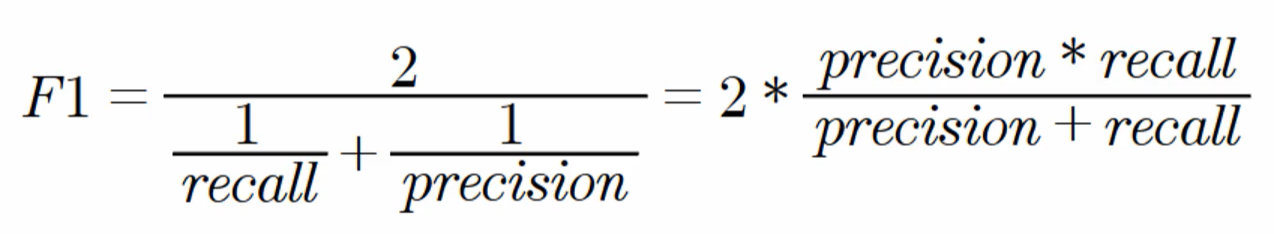
- 정밀도가 0.9, 재현율이 0.1일 경우 -> F1 스코어는 0.18
- 정밀도가 0.5, 재현율이 0.5일 경우 -> F1 스코어는 0.50
- 사이킷런에서 f1_score() 함수 제공
from sklearn.metrics import f1_score
# pred는 위에서 구했다고 치자
f1 = f1_score(y_test , pred)
print('F1 스코어: {0:.4f}'.format(f1))결과
F1 스코어: 0.7966
-----
def get_clf_eval(y_test , pred):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
# F1 스코어 추가
f1 = f1_score(y_test,pred)
print('오차 행렬')
print(confusion)
# f1 score print 추가
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f}, F1:{3:.4f}'.format(accuracy, precision, recall, f1))
thresholds = [0.4 , 0.45 , 0.50 , 0.55 , 0.60]
pred_proba = lr_clf.predict_proba(X_test)
get_eval_by_threshold(y_test, pred_proba[:,1].reshape(-1,1), thresholds)결과
임곗값: 0.4
오차 행렬
[[97 21]
[11 50]]
정확도: 0.8212, 정밀도: 0.7042, 재현율: 0.8197, F1:0.7576
임곗값: 0.45
오차 행렬
[[105 13]
[ 13 48]]
정확도: 0.8547, 정밀도: 0.7869, 재현율: 0.7869, F1:0.7869
임곗값: 0.5
오차 행렬
[[108 10]
[ 14 47]]
정확도: 0.8659, 정밀도: 0.8246, 재현율: 0.7705, F1:0.7966
임곗값: 0.55
오차 행렬
[[111 7]
[ 16 45]]
정확도: 0.8715, 정밀도: 0.8654, 재현율: 0.7377, F1:0.7965
임곗값: 0.6
오차 행렬
[[113 5]
[ 17 44]]
정확도: 0.8771, 정밀도: 0.8980, 재현율: 0.7213, F1:0.8000
ROC 곡선 (Receiver Operation Characteristic Curve)
- FPR이 변할 때 TPR이 어떻게 변하는지를 나타내는 곡선
- 아래의 그래프와 같이 둘의 관계는 곡선의 평태로 나타남
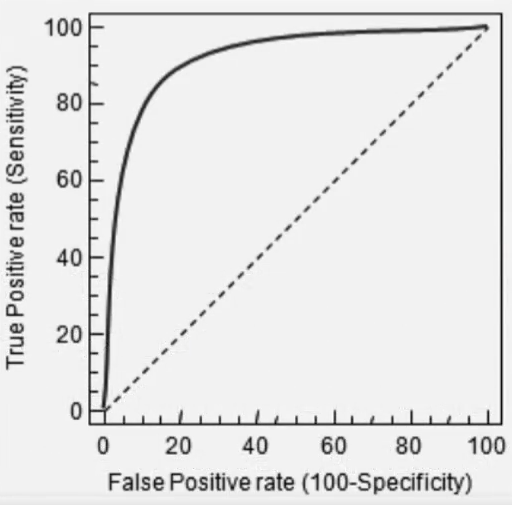
- TPR (True Positive Rate): 재현율!(=민감도) -> TP / (FN + TP)
- FPR (False Positive Rate): 실제 Negative를 Positive로 잘못 예측한 비율 -> FP / (FP + TN)
→ 임곗값을 1로 하면 FPR은 0 / 임곗값을 0으로 하면 FPR은 1
→ FPR을 조금씩 줄여간다고 생각하고 그래프 관찰하기!
→ FPR의 감소 비율에 비해 TPR(Recall)의 감소 비율이 더 작아야 (== 값이 높아진 상태로 오래 유지되어야) 좋음
AUC (Area Under Curve)
- ROC 곡선 밑의 면적을 구함
- 1에 가까울수록 좋은 수치임!
사이킷런 함수

ROC AUC 실습
from sklearn.metrics import roc_curve
# 레이블 값이 1일때의 예측 확률을 추출
pred_proba_class1 = lr_clf.predict_proba(X_test)[:, 1]
fprs , tprs , thresholds = roc_curve(y_test, pred_proba_class1)
# 반환된 임곗값 배열에서 샘플로 데이터를 추출하되, 임곗값을 5 Step으로 추출.
# thresholds[0]은 max(예측확률)+1로 임의 설정됨. 이를 제외하기 위해 np.arange는 1부터 시작
thr_index = np.arange(1, thresholds.shape[0], 5)
print('샘플 추출을 위한 임곗값 배열의 index:', thr_index)
print('샘플 index로 추출한 임곗값: ', np.round(thresholds[thr_index], 2))
# 5 step 단위로 추출된 임계값에 따른 FPR, TPR 값
print('샘플 임곗값별 FPR: ', np.round(fprs[thr_index], 3))
print('샘플 임곗값별 TPR: ', np.round(tprs[thr_index], 3))결과
샘플 추출을 위한 임곗값 배열의 index: [ 1 6 11 16 21 26 31 36 41 46]
샘플 index로 추출한 임곗값: [0.94 0.73 0.62 0.52 0.44 0.28 0.15 0.14 0.13 0.12]
샘플 임곗값별 FPR: [0. 0.008 0.025 0.076 0.127 0.254 0.576 0.61 0.746 0.847]
샘플 임곗값별 TPR: [0.016 0.492 0.705 0.738 0.803 0.885 0.902 0.951 0.967 1. ]
-----
def roc_curve_plot(y_test , pred_proba_c1):
# 임곗값에 따른 FPR, TPR 값을 반환 받음.
fprs , tprs , thresholds = roc_curve(y_test ,pred_proba_c1)
# ROC Curve를 plot 곡선으로 그림.
plt.plot(fprs , tprs, label='ROC')
# 가운데 대각선 직선을 그림.
plt.plot([0, 1], [0, 1], 'k--', label='Random')
# FPR X 축의 Scale을 0.1 단위로 변경, X,Y 축명 설정등
start, end = plt.xlim()
plt.xticks(np.round(np.arange(start, end, 0.1),2))
plt.xlim(0,1); plt.ylim(0,1)
plt.xlabel('FPR( 1 - Sensitivity )'); plt.ylabel('TPR( Recall )')
plt.legend()
plt.show()
roc_curve_plot(y_test, lr_clf.predict_proba(X_test)[:, 1] )결과

-----
from sklearn.metrics import roc_auc_score
pred_proba = lr_clf.predict_proba(X_test)[:, 1]
roc_score = roc_auc_score(y_test, pred_proba)
print('ROC AUC 값: {0:.4f}'.format(roc_score))결과
ROC AUC 값: 0.8987
-----
-> get_clf_eval 함수에 ROC-AUC 추가
def get_clf_eval(y_test, pred=None, pred_proba=None):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
f1 = f1_score(y_test,pred)
# ROC-AUC 추가
roc_auc = roc_auc_score(y_test, pred_proba)
print('오차 행렬')
print(confusion)
# ROC-AUC print 추가
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f},\
F1: {3:.4f}, AUC:{4:.4f}'.format(accuracy, precision, recall, f1, roc_auc))
평가 실습 - 피마 인디언 당뇨병 예측
피마 인디언 당뇨병 데이터셋
- 피마라는 지역에 서구 문명이 들어오면서, 식습관 음식 등의 변화로 인해 당뇨병이 유행하게 됨
(jupyter notebook 3장 3.6 피마 인디언 당뇨병 예측)
'프로그래밍 언어 > python' 카테고리의 다른 글
| 앙상블 학습, 하이퍼 파라미터 튜닝 | 4주차 | 파이썬 머신러닝 완벽가이드 (0) | 2023.02.20 |
|---|---|
| 분류, 결정트리, Overfitting | 3주차 - 5 | 파이썬 머신러닝 완벽가이드 (0) | 2023.01.23 |
| 사이킷런, Iris data_K Fold 검증, 데이터 인코딩, 스케일링 | 3주차 - 1, 2 | 파이썬 머신러닝 완벽가이드 (0) | 2023.01.21 |
| 데이터 시각화(Matplotlib, Seaborn) | 2주차 | 파이썬 머신러닝 완벽가이드 (0) | 2023.01.08 |
| pandas 알아보기 | 1주차 - 2 | 파이썬 머신러닝 완벽가이드 (0) | 2023.01.05 |




